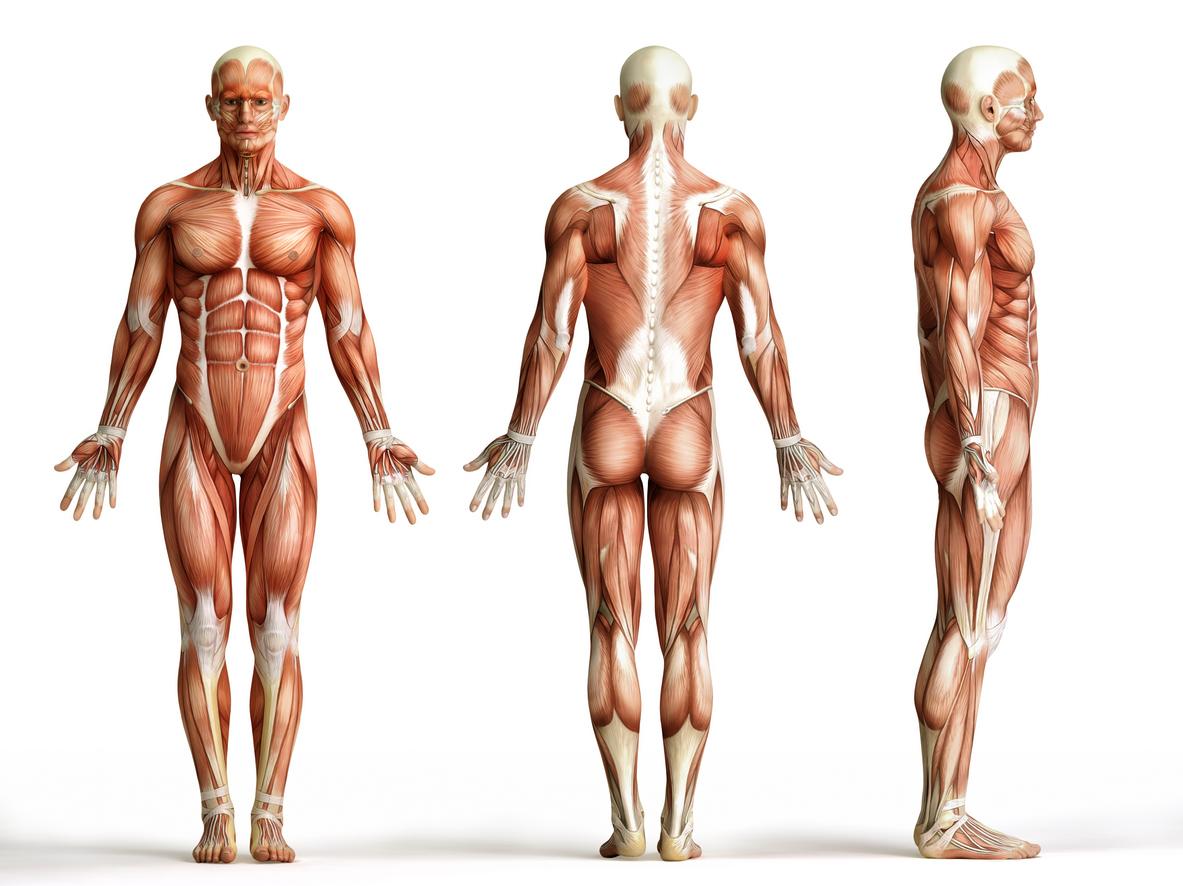
- Les scientifiques sont désormais capables de prédire la structure de 350 000 protéines, contre 170 000 auparavant.
- Pour cela, ils ont utilisé un outil d’intelligence artificielle pouvant reconnaître les acides aminés qui composent les protéines pour modéliser leur forme en 3D.
Des scientifiques ont réussi à mettre au point une base de données regroupant les structures de 350 000 protéines de l’organisme humain et 20 protéines d’autres organismes, comme la levure. Cette prouesse a été possible grâce à un outil d’intelligence artificielle nommé AlphaFold, dont l’algorithme a été entraîné à reconnaître les acides aminés qui constituent la structure des protéines. Pour cela, les scientifiques se sont servis des données des 170 000 structures protéiques qu’ils connaissaient déjà. Leurs travaux ont été publiés dans la revue Nature.
Comprendre les acides aminés pour prédire la structure des protéines
Pour comprendre l’intérêt de cette découverte, il faut au préalable bien en définir les termes. Les protéines, tout d’abord, sont les principales composantes des structures de toutes les cellules du corps humain. Elles donnent des instructions aux cellules afin que celles-ci remplissent leurs fonctions. L’ensemble des protéines d’un organisme s’appelle le protéome. Sa composition se modifie en fonction des conditions environnementales internes mais aussi externes aux cellules. L’intérêt de l’étude du protéome - et donc les milliers de protéines qui composent l’organisme - est de mieux comprendre les mécanismes du vivant et de notre corps.
Mais pour analyser les protéines, il faut étudier leurs acides aminés. Il s’agit de molécules unitaires qui vont donner une forme aux protéines en les repliant. Chaque protéine repliée dispose d’une configuration spécifique qui lui donne une forme distincte de celle des autres protéines. Ces repliements créent donc une large diversité de protéines. Pour les scientifiques, l’enjeu est de pouvoir prédire ces repliements afin d’anticiper la structure que les protéines peuvent avoir. Jusqu’à AlphaFold, seules 170 000 structures de protéines étaient connues.
Mieux connaître les protéines pour créer des médicaments
AlphaFold sait reconnaître 60% acides aminés de l’organisme, mais cela ne suffisait pas pour prédire les protéines : il fallait aussi modéliser leur structure en 3D, ce qui permet de prédire la forme qu’elles pourraient prendre. Autrement dit, imaginer comment elles vont être repliées par les acides aminés car c’est cette action de repliement qui définit la ou les fonctions que la protéine va remplir. Ainsi, cet outil d’intelligence artificielle a réussi à prédire la forme de 350 000 protéines, soit 44% de celles humaines.
“Ce qui nous a pris des mois et des années à faire, AlphaFold a pu le faire en un week-end, explique John McGeehan, dans une série de commentaires indépendants dédiés à cette étude. J’ai le sentiment que nous venons de faire un bond d’au moins un an par rapport à la situation d’hier”. En effet, une fois l’algorithme d’intelligence artificielle mis au point, l’analyse est très rapide. D’autre part, cet outil d’intelligence artificielle a aussi mis en avant l’existence de nombreuses protéines humaines désordonnées, ce qui signifie que l’algorithme n'a pas réussi à en prédire la structure.
Les auteurs concluent que ces protéines n'adoptent pas une forme unique, mais variable. Le premier intérêt de ce catalogue de 350 000 protéines est d’améliorer les connaissances fondamentales sur la biologie humaine, végétale et animale. Tous les scientifiques peuvent par exemple s’y référer pour créer de nouveaux médicaments, car certaines protéines résistent ou participent à une infection. Actuellement, une équipe de chercheurs utilisent AlphaFold pour analyser la façon dont les protéines du coronavirus s’attachent aux cellules humaines.